주제가 매우 좁은 영역인 경우 탐색이 비교적 수월할 수 있습니다.
하지만 뭔가 특정 주제에 해당하는 연구를 가급적 빠뜨림 없이 폭넓게 찾고 싶을 때에는 단순히 google scholar, pubmed에서 보면서 괜찮네? 이것은 따로 링크를 타고 들어가서 원문을 보고 저장하고 싶은 경우 하나하나 endnote나 zotero에 넣는 과정은 다소 번거로울 수 있습니다.
이번 글에서는 지금까지 제가 찾은 툴들을 가급적 다 사용해보면서 사용 사례를 기술해보고 효과적이면서 효율적인 문헌탐색을 나름대로 정리해보고 자 합니다. 물론 체계적 문헌탐색 기법이 있지만 이 분야도 전문적 영역이라 그러한 체계적 문헌 리뷰를 해보지 않는 저로서는 그러한 학술적 접근보다 일반인 수준에서 가급적 폭넓고 유용한 툴을 어떻게 사용하는가 하는 관점에서 접근하고자 합니다.
문헌 탐색 과정 정리
pubmed 검색 - > Rayyan에 업로드하고 스크리닝 시작 -> Connected Papers 또는 Research Rabbit으로 문헌 확장 > Zotero로 논문 정리 > zetero의 문헌들에서 필요한 내용을 탐색하여 정리한다.
(업데이트) demension.ai를 이용하여 문헌/펀드/임상시험/특허 한번에 찾고, 정리가 가능
1. 시작: pubmed 검색 또는 Research Rabbit을 이용한 연관 검색
PubMed 웹 검색 vs. PubMed API: 차이점 간단 비교
- PubMed 웹 검색:
- 장점: 직관적이고, 계정 없이 바로 사용 가능, 소규모 검색에 적합.
- 단점: 대량 데이터 수집 시 수동 작업 필요, 자동화 불가, 결과 저장 방식의 한계.
- PubMed API:
- 장점: 대량 데이터 자동화, 정형화된 데이터 형식, 반복 작업 효율화.
- 단점: 프로그래밍 지식 필요, 초기 설정 비용(시간/학습).
PubMed API를 사용하는 장점
Transfusion RAG-LLM 구축을 위한 문헌 탐색에서 PubMed API가 웹 검색보다 유리한 이유는 다음과 같습니다:
- 대량 데이터의 효율적 수집
- 웹 검색의 한계: PubMed 웹사이트에서 검색하면 한 번에 표시되는 결과는 20~200개 정도로 제한되며, 페이지별로 수동으로 이동해 RIS 파일로 내보내야 합니다. 예를 들어, "transfusion medicine" AND "large language models"로 검색 시 수백 개 결과가 나오면 이를 모두 저장하려면 시간이 많이 걸립니다.
- API 장점: PubMed API(특히 E-Utilities)를 사용하면 검색 결과를 한 번에 수천 개까지 자동으로 가져올 수 있습니다. 이를 Python 스크립트로 실행하면 수동 클릭 없이 모든 논문을 빠르게 수집 가능합니다.
- 실행 예시: SurgeryLLM 논문(https://doi.org/10.1038/s41746-024-01391-3)에서 외부 가이드라인(ACC/AHA 등)을 대량으로 통합한 사례를 참고하면, API로 문헌을 체계적으로 수집하는 것이 데이터 준비에 유리합니다.
- 정형화된 데이터 제공
- 웹 검색의 한계: 웹에서 내보낸 RIS 파일은 제목, 초록, 저자 등 기본 정보만 포함하며, 추가 메타데이터(예: MeSH 용어, 출판 유형)를 얻으려면 개별 논문을 열어야 합니다.
- API 장점: API는 XML 또는 JSON 형식으로 논문의 상세 메타데이터(예: MeSH 주제어, 키워드, DOI, PMID)를 제공합니다. 이는 Transfusion RAG-LLM의 지식 베이스를 구축할 때 주제별 분류(예: "blood transfusion", "apheresis")를 자동화하는 데 유용합니다.
- 활용 예시: "transfusion medicine" 관련 논문을 MeSH 태그로 필터링해 수혈 부작용(TRALI 등)에 초점을 맞춘 논문만 추출 가능.
- 자동화 및 재현 가능성
- 웹 검색의 한계: 검색어를 입력하고 결과를 저장하는 과정은 수동적이어서, 나중에 동일한 검색을 반복하려면 기록을 꼼꼼히 남겨야 합니다.
- API 장점: Python이나 R로 작성한 API 스크립트를 사용하면 검색 조건(키워드, 날짜 범위 등)을 코드로 저장해 언제든 동일한 결과를 재현할 수 있습니다. 이는 연구의 투명성과 재현성을 높이는 데 필수적입니다(SurgeryLLM 논문에서 코드 공유 정책을 언급한 점 참고).
- 실행 예시: SurgeryLLM처럼 대규모 데이터셋 구축 시, API로 매일 업데이트되는 논문을 주기적으로 수집 가능.
- 대규모 스크리닝 및 통합 작업 지원
- 웹 검색의 한계: 수백~수천 개 논문을 Rayyan에 업로드하려면 웹에서 내보낸 파일을 하나씩 정리해야 하며, 이 과정에서 누락 위험이 있습니다.
- API 장점: API로 수집한 데이터를 바로 Rayyan, Zotero와 통합할 수 있도록 스크립트를 작성하면 워크플로우가 간소화됩니다. 예를 들어, PMID 목록을 CSV로 저장한 뒤 Rayyan에 직접 업로드 가능.
- 활용 예시: Transfusion RAG-LLM에 필요한 논문(예: RAG와 수혈 관련)을 한 번에 수집해 Rayyan으로 스크리닝.
- 시간 절약 및 확장성
- 웹 검색의 한계: 논문 수가 많아질수록 수동 검색과 저장의 비효율성이 커집니다.
- API 장점: 초기 설정 후에는 검색, 다운로드, 저장이 자동화되어 수십~수백 시간 절약 가능. 이후 Apheresis RAG-LLM 등 다른 주제로 확장 시에도 동일 스크립트를 재사용 가능.
- 참고: SurgeryLLM 논문은 LangChain과 ChromaDB로 외부 지식을 통합했는데, API로 수집된 데이터는 이런 시스템에 바로 활용될 수 있는 구조화된 형태로 제공됩니다.
PubMed API 사용의 단점
- 학습 곡선: Python이나 API 사용 경험이 없으면 초기 설정(예: E-Utilities 학습)에 시간이 걸립니다.
- 속도 제한: PubMed API는 무료 사용 시 초당 3건 요청 제한이 있으며, 대량 요청 시 API 키를 신청해야 합니다.
- 복잡성: 단순 검색보다 설정이 복잡할 수 있음.
PubMed 웹 검색만으로 충분한 경우
- 논문 수가 적거나(예: 50개 미만), 일회성 탐색이라면 웹 검색 후 RIS 파일로 내보내고 Rayyan/Zotero로 관리하는 방식이 빠르고 간단합니다.
- 프로그래밍에 익숙하지 않다면 웹 검색이 더 접근하기 쉬울 수 있습니다.
실제로 따라 할 수 있는 제안 (API vs. 웹 선택)
선택 1: PubMed API 사용
- 준비:
- Python 설치(3.9 이상, SurgeryLLM에서 사용된 버전 참고).
- biopython 라이브러리 설치: pip install biopython.
- 스크립트 작성:
python
WrapCopy
from Bio import Entrez Entrez.email = "your.email@example.com" # NCBI 요구 사항 search_term = '"Retrieval-Augmented Generation" AND "transfusion medicine"' handle = Entrez.esearch(db="pubmed", term=search_term, retmax=100) record = Entrez.read(handle) pmids = record["IdList"] print(f"Found {len(pmids)} articles: {pmids}") - 데이터 가져오기:
- PMID로 논문 메타데이터를 XML로 다운로드하고, 필요 시 PDF 링크를 추출.
- 통합:
- 결과를 CSV로 저장 후 Rayyan에 업로드.
장점: 대량 논문(100개 이상)을 한 번에 처리 가능.
선택 2: PubMed 웹 검색
- 검색:
- PubMed 웹사이트(pubmed.ncbi.nlm.nih.gov)에 접속.
- 검색어 입력: "Retrieval-Augmented Generation" AND "transfusion medicine".
- 내보내기:
- 결과 페이지에서 "Send to" → "File" → "Format: RIS" 선택 후 다운로드.
- 관리:
- RIS 파일을 Rayyan에 업로드하고 스크리닝 시작.
장점: 설정 없이 바로 시작 가능, 소규모 작업에 적합.
결론: 어떤 방식을 추천하나?
- 소규모 탐색(50개 이하 논문): PubMed 웹 검색으로 충분합니다. RIS 파일을 다운로드해 Rayyan에서 바로 스크리닝하세요.
- 대규모, 장기 프로젝트: PubMed API를 추천합니다. 외부 지식을 대량으로 통합하려면 API의 자동화와 정형화된 데이터가 필수적입니다. 초기 학습 비용은 있지만 장기적으로 시간과 노력을 절약하며, Research Rabbit이나 Zotero와의 통합도 더 원활합니다.
2. rayyan.ai 업로드

좋은 점은 혼자 또는 여럿이서 공동으로 문헌을 선별할 수 있게되어 있고, 사유도 적을 수 있습니다. 제외된 문헌들도 유지되기 때문에 언제든 재분류가 가능합니다.
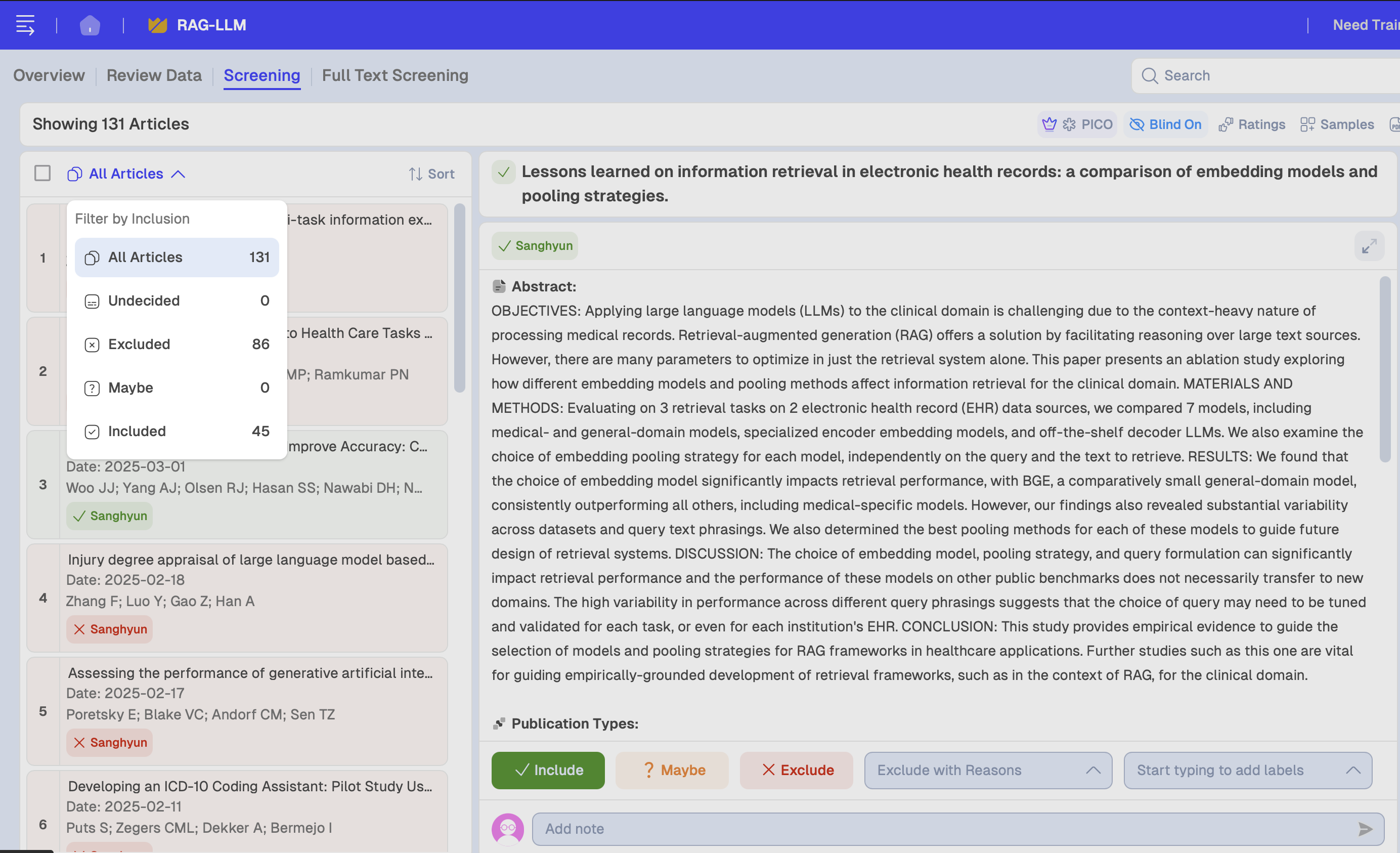
131개의 문헌 중 45편을 포함하였고, 나머지 88편은 제외하였습니다.
이제 이를 zotero에 옮기겠습니다.
3. Zotero에 업로드
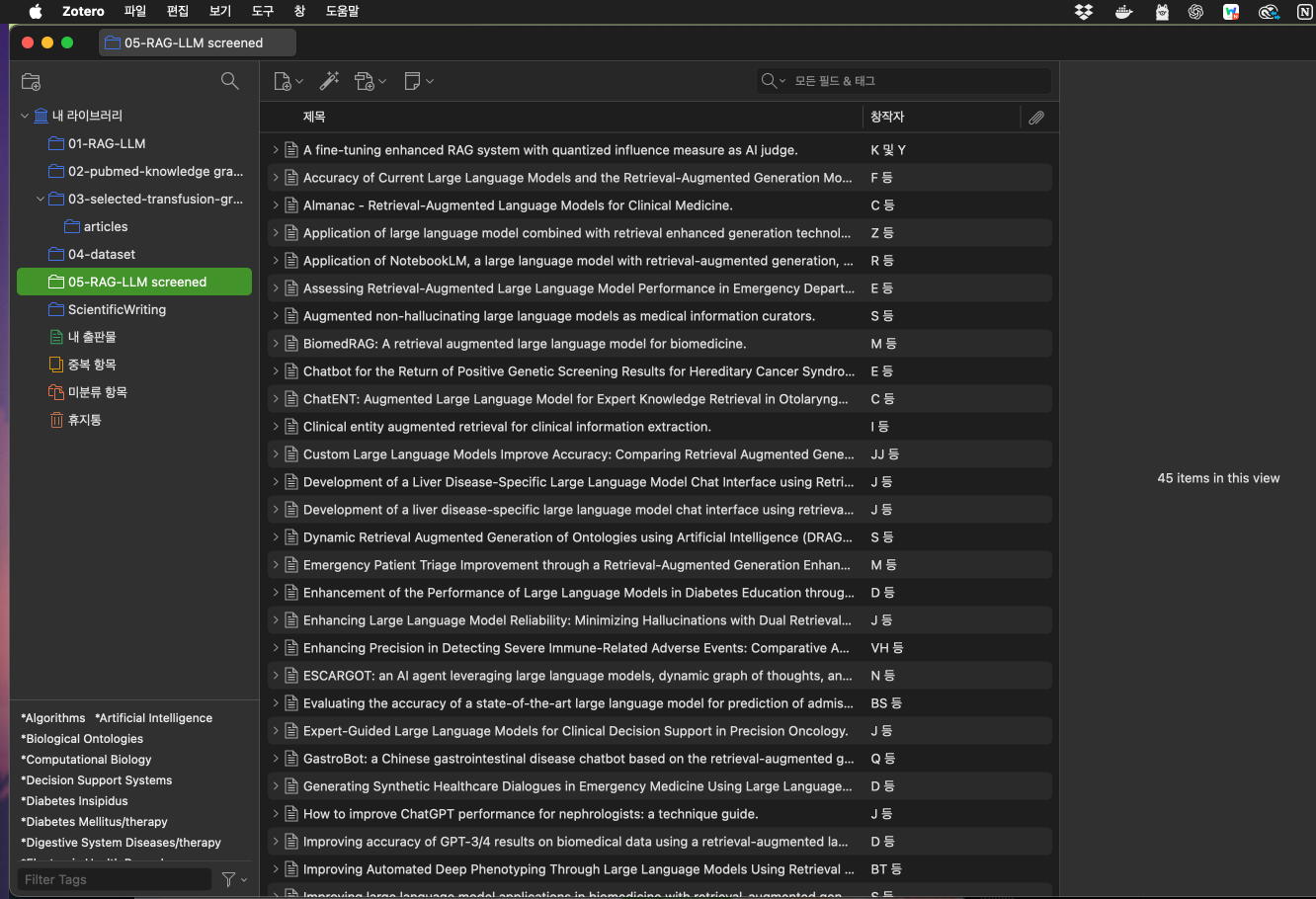
다음과 같이 선별된 43개의 문헌이 잘 업로드 되었습니다.
이제 이들 키 저널을 하나하나 원문을 보면서 핵심 저널을 찾고 그 저널과 연결된 연구들은 research rabbit으로 쉽게 찾을 수 있습니다.
4. 연관 문헌 탐색: connected papers 또는 research rabbit을 이용
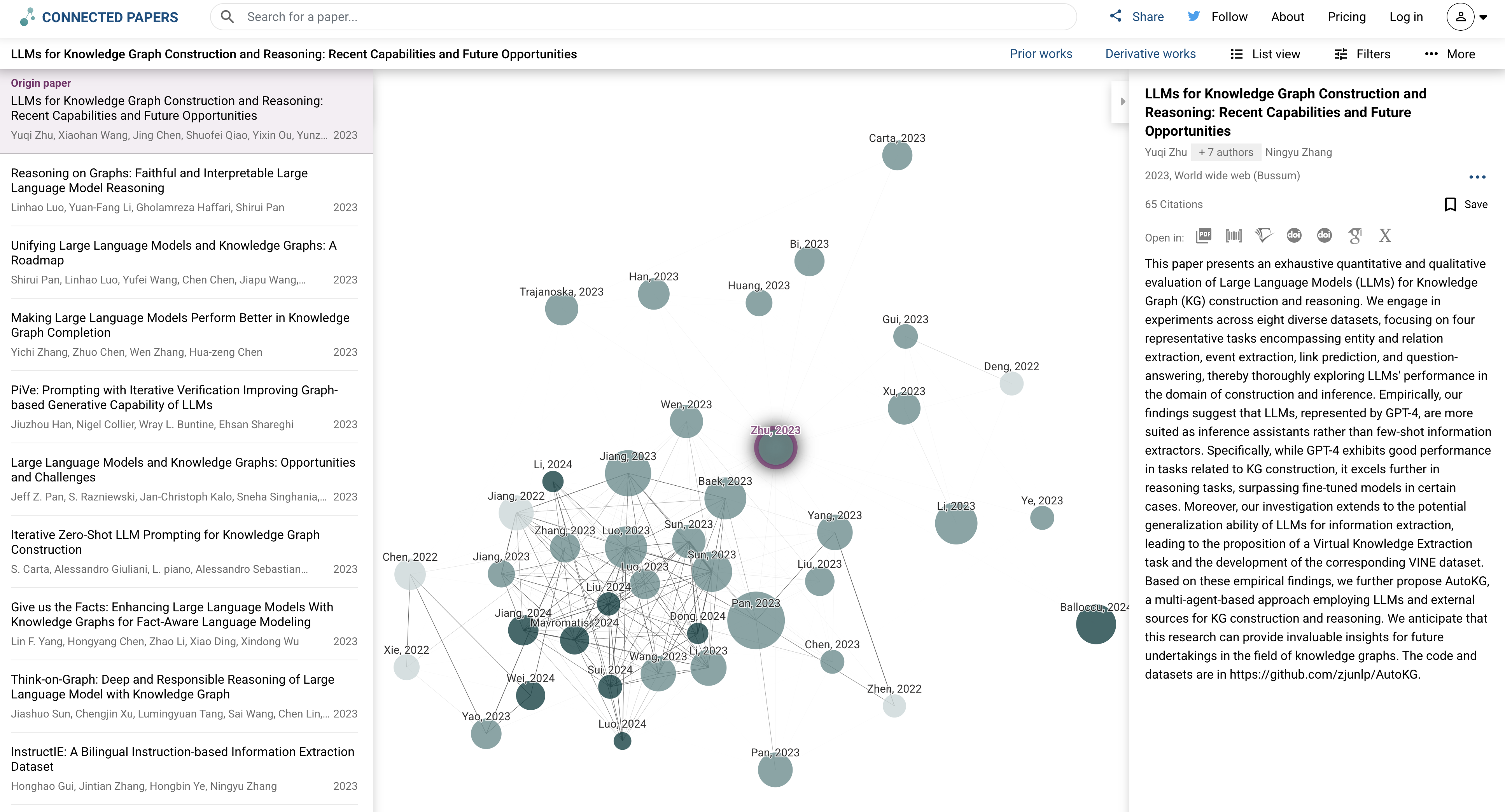
특정 문헌을 중심으로 연관된 문헌을 쉽게 탐색하여 추가적인 문헌을 탐색할 수 있다.
개인적으로는 research rabbit보다는 conneted papers가 더 좋은 것 같다는 생각이 듭니다. 아직 research rabbit에 대해 경험이 많지 않아서인지도 모르겠습니다. conneted papers는 직관적으로 접근이 가능해서 편하다는 느낌이 들었습니다. 원의 크기는 인용횟수의 크기를 나타내서 영향력이 큰 논문을 쉽게 찾을 수 있어 정말 좋았습니다. conneted papers를 이용하는 가장 큰 이유중의 하나입니다.
'의학' 카테고리의 다른 글
| 눈에 띄는 AI 에이전트 시리즈(2); Designed For Academic Writing (0) | 2025.01.08 |
|---|---|
| AI의 의학도전 (12) - 대규모 언어 모델의 이점과 한계: 혈액학 분야에서의 활용 (0) | 2024.11.22 |
| AI의 의학도전 (11) - "전문의급 실력" GPT-4의 안과 진단 능력 첫 검증: 422개 임상 사례 분석 결과 (2) | 2024.11.21 |
| 면역치료의 양날의 검: CRS의 모든 것 (2) - 패혈증과의 감별 마커 (0) | 2024.11.18 |
| B-급성 림프모구 백혈병(B-ALL)에서 CRLF2의 면역표현형 마커로서의 연관성과 유용성 (0) | 2024.11.15 |